|
|
新京报快讯(记者 李玉坤)《国家科学评论》3月3日在线发布了北大生命科学院研究员陆剑等人的论文,媒体对此论文解读称,新冠病毒已经突变,演化为两个亚型,其中一个亚型“传染性”更强(aggressive)。
# \! u( r7 R8 |. B1 v! x! K- ? 3月11日晚,记者发现陆剑等人已经重新提交了手稿,在文末解释称,“aggressive”(具有侵略性)用词具有误导性,他们没有关于病毒致病性的结论。
* r; u4 G, i! i. c4 K0 }新冠病毒变异后会更有“侵略性”?
6 b& W- u# K6 r/ E' x% n3 X 论文作者补充声明
% m6 q2 S; V$ O( ~3 s* P+ q& V 陆剑等人研究了103个新冠病毒基因组发现,由于一个蛋白编码位点的突变产生的差别,将新冠病毒毒株清晰分为两组,被媒体称为“亚型”。但有学者认为,目前变异造成的差异不足以叫“亚型”,叫“分枝”(clade)比较合适。
$ S7 n$ V2 j. J! o( E" e+ R 陆剑等人在重新提交的论文中,做了补充性声明:$ ]2 u8 S! Y0 {4 t, B. N
“在我们最近发表的文章,分析结果显示,103个SARS-CoV-2病毒基因组存在两种不同的谱系;分别称之为‘L’和‘S’谱系。我们用来定义L和S谱系的氨基酸位点位于ORF8(开放阅读框8。所谓开放阅读框是指是DNA序列中具有编码蛋白质潜能的序列——记者注。)基因,这个基因还没有发现具有任何已知的重要功能。基于‘L’谱系的频率高于谱系S的发现,我们将L谱系描述为‘aggressive’(具有侵略性)。我们现在认识到,在本研究阐述的内容中,‘侵略性’一词会具有误导性,应该用更精确的术语‘更高的频率’代替。简而言之,尽管我们已经发现这两个谱系自然并存,但我们没有提供任何证据支持关于SARS-CoV-2毒力或致病性的任何流行病学结论。因此,我们将在本文的印刷版本中进行更正,以避免产生误导。”
4 f. `0 W# t) y& z0 r 在此前的报道中,媒体多用“亚型”这一表述。从声明中可以看出,陆剑等人并不认为突变产生了两个“亚型”,而是用了“谱系”这一说法。
1 p( h. J. g# _8 I 在论文刚公布的时候,媒体报道,“新变异出的L亚型更具传染性”。对于这个观点,陆剑等人做了重点更正,认为此前用词具有误导性。2 s6 w( P3 C" @; F9 d- U4 @) o( t9 d
新冠病毒有多爱突变?8 ^& l) V. S) j. W' P
已记录481个变异8 ]! ~/ J3 D" G' c! J
湖北省疾控中心卫生检测检验研究所流感参比实验室主任刘琳琳等人在另一篇论文中提到,根据数据库,新冠病毒的基因组大小从29,409bp到29,911bp不等,包括183个可变位点。
& z0 U4 d ^+ b# f6 {. X0 S 记者发现,截至3月12日5时,国家生物信息中心2019新型冠状病毒信息库中已经记录了482个新冠病毒的变异。
7 t2 i5 P. u0 O+ |; }* j! w g9 d5 G新型冠状病毒信息库记录4种变异类型,分别是SNP(单核苷酸多性态,就是单个碱基的改变)、deletation(缺失,一个或者多个核苷酸丢失)、insertion(插入,多了一个或多个核苷酸)、indel(插入或缺失)。其中SNP最多,有461条。其次是deletation,有20条。因为新冠病毒是RNA单链,这种病毒SNP变异特别常见。
: m4 k# P! j, w! q6 p2 C- N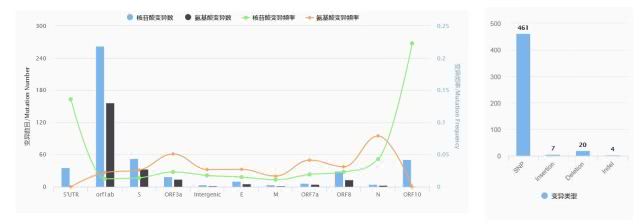 , j9 [% w3 \2 C/ u8 \; C- V* A , j9 [% w3 \2 C/ u8 \; C- V* A
% b2 M$ Q+ |- x+ v+ E 新冠病毒各基因的变异情况。
; i$ r3 S: w8 J+ J) x 陆剑等人用来定义L和S谱系的氨基酸位点位于ORF8(开放阅读框8)。从新型冠状病毒信息库的数据来看,ORF8的变异不是最多的。并且,陆剑等人表示这个基因还没有发现具有任何已知的重要功能。+ O4 R* r6 ^) o
记者了解到,S基因具有重要功能,这个基因用来生产S蛋白(spikeprotein,翻译为棘突蛋白、长钉蛋白、穗蛋白),棘突蛋白会和病毒宿主细胞受体ACE2结合,使得病毒能够侵入到细胞内。
6 ]# i7 V: @! M" u7 O 在S基因中,有一处重要突变一直是各国科学家研究的重点。南开大学高山等人发现,新冠病毒的受体结合基序中多了一个Furin蛋白酶切位点,这在蝙蝠冠状病毒和穿山甲冠状病毒中都没有发现。不同于SARS等其他大部分Beta冠状病毒,这个变异使得新冠病毒可能采用了HIV等其他病毒的类似的包装机制,它的棘突蛋白获得了更高的侵染细胞的效率,更具感染性。
% O( a7 d0 M5 F' S& J9 B1 X 两个谱系有何差别?
0 e' O2 R. X6 U L型由S型衍化而来
# `1 n) J8 y2 s" z: z/ e 陆剑等人用完全连锁的SNP区分了新冠病毒的两种谱系,在103株新冠病毒病毒株中,有101株表现出两个SNP之间的完全连锁:72株表现出“CT”单倍型(定义为“L”型,因为是亮氨酸的密码子),29株在这两个位点表现出“TC”单倍型(定义为“S”型,因为是丝氨酸的密码子)。由此可见,L为主要类型(占70%),S为次要类型(占30%)。; {4 x1 }# [9 r4 V9 ?$ |
他们将新冠病毒和其他高度相关的病毒基因组比对,发现S谱系的核苷酸与最密切相关的病毒的同源位点相同,L型比S型更多,但S型实际上更“老”。不过,尽管L型是从古代S型新进化而来的,但它在人类群体中的传播或复制速度更快,从而导致其积累的突变比S型更多。
7 b+ | Z! p- `- ]; n; R 从武汉分离出的27种病毒中,有26种(96.3%)为L型,只有1种(3.7%)为S型。但是,在武汉以外地区分离出的其他73种病毒中,L型为45种(61.6%),S型为28种(38.4%)。这种比较表明,L型在武汉比其他地方普遍得多。
9 q- O) j$ e0 E7 _0 Y: \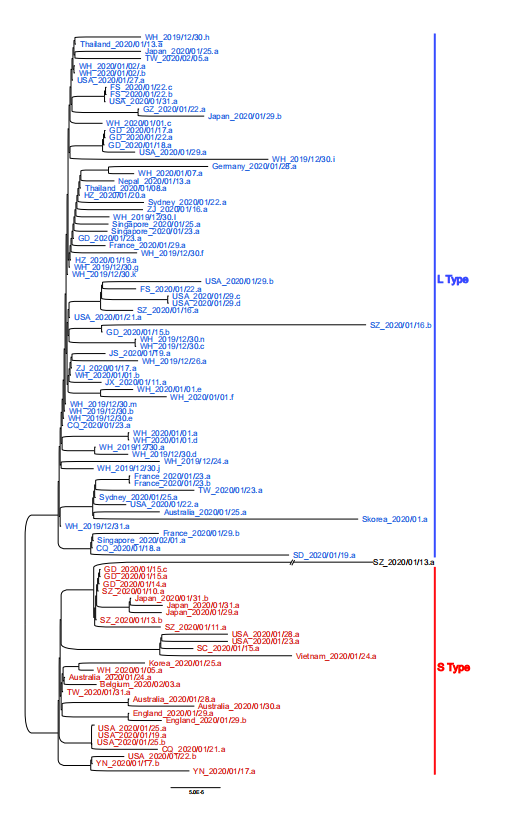
' z- ^( ~1 r1 `/ n! o. U% k
, y5 q* h I1 Z+ E6 W3 i 新冠病毒分化的两个谱系。& y# V" J4 d. r
刘琳琳等人做的研究,也显示出了类似的进化和分布关系。. _" I4 G( b& I7 n" Q
他们通过分析146个基因组,发现了183个替换位点,将新冠病毒分为80个单倍型。其中,与其祖先单倍型相比,一个被称为H3的单倍型及其衍生的单倍型数量很大,并在湖北(尤其武汉)广泛分布。除中国外,其他15个国家提供的65个病毒样本被分配给43个单倍型。其中,H3衍生出来的单倍型有27种,它的两个祖先单倍型H14衍生的单倍型11种,H15衍生的单倍型3种。( Z H, X( \4 ?
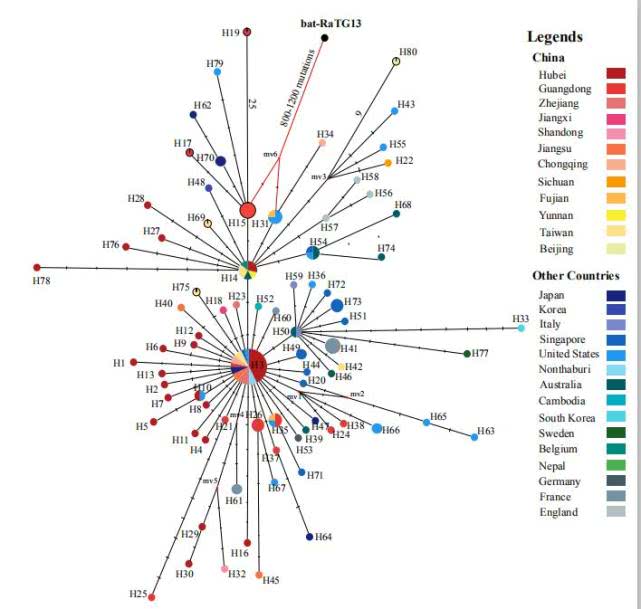
2 x1 b: x: H$ F- `! T6 L1 x+ [/ f
) K0 |- p, @! f% T8 e" O 各单倍型之间的衍化关系。0 N* O; { {: K' U
值得一提的是,陆剑等人发现,有两个样本包含了S和L两种类型,这两个样本都来自国外——美国和澳大利亚。他们猜测,患者可能感染了至少两种不同的新冠病毒。8 q/ U) `1 j. H; `$ j7 n- S
直接来源到底是哪种动物?( n) e- Y$ a: j' ]
穿山甲不太可能1 l9 l% ]: S2 ~2 k
陆剑等人除了研究新冠病毒的进化外,还研究了新冠病毒的起源,着重分析了是来自蝙蝠还是穿山甲。 r9 U& g7 W# ]
全基因组系统发生树表明新冠病毒最接近蝙蝠冠状病毒(RaTG13),其次是穿山甲相关冠状病毒,然后是人的SARS病毒。刘琳琳等人也得出了相同的结论,基于系统发生树,蝙蝠冠状病毒(bat-RaTG13-CoV)与新冠病毒和两个从广东采集的穿山甲冠状病毒的姊妹病毒的遗传距离相对较大,穿山甲可能不是新冠病毒中间宿主。( a6 v# K" `2 D3 A* S, \3 R
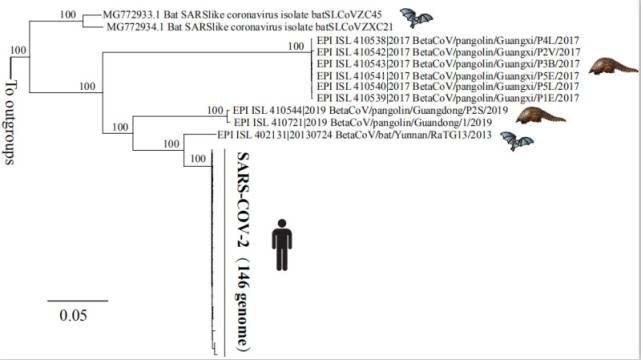
: ?$ ^) P* }: e& I, z& z! Q* W6 y9 i J7 O; p
蝙蝠、穿山甲和人类新冠病毒的遗传距离。5 ?5 X6 l2 S6 | [5 e) u. t; t
不过,陆剑等人发现,尽管新冠病毒与蝙蝠SARS相关冠状病毒(SARSr-CoV-RaTG13)之间的基因组核苷酸差异仅4%,但中性位点的差异为17%,表明两种病毒之间的差异比以前估计的要大要多。作者做了一个对比,这个差异是人与黑猩猩之间差距的14倍,是人与恒河猴之间差距的2倍。' u! t8 t% U8 s
此外,有一些研究表明穿山甲可能为新冠病毒提供了部分棘突基因(上文说的S基因)。棘突蛋白受体结合域中的6个关键氨基酸残基,和SARS病毒相比,新冠病毒有5个残基都不同,并且新冠病毒与受体ACE2的结合亲和力更高。蝙蝠冠状病毒(RaTG13)也是6个中只有1个相同,但有趣的是,穿山甲冠状病毒和新冠病毒的6个关键氨基酸残基是相同的。4 }3 o m( {2 `1 Z: O' P5 @7 e! l* z8 B
有人提出,新冠病毒的棘突蛋白受体结合域可能是由穿山甲中最近的重组事件引起的。陆剑等人认为,尽管棘突蛋白产生过重组,但新冠病毒和穿山甲冠状病毒中相同的功能位点也可能是巧合的趋同进化的结果,香港大学管轶等人在此前的论文中表达了相同的观点。
5 ?4 u, X# i- w/ [/ L8 }" z. \5 ` 【主要参考论文】* L7 D0 w a9 f2 T I# d: n
Genome-widedata inferring the evolution and population demography of the novel pneumonia coronavirus (SARS-CoV-2). bioRxiv2020.03.04.976662! x$ e4 E, H, Q7 o; i4 L/ n- J
On the origin and continuing evolution of SARS-CoV-2. National Science Review, nwaa036
6 H2 i k5 s$ N: v) h 新京报记者 李玉坤, t1 j+ H/ ^+ o8 P" C1 `
y8 o0 R; \9 l1 ]5 U
. P& \. j, @* \0 X9 J7 g' |6 E7 C2 c2 h2 D
; h& Q; H* K8 v. T
/ Z' \: ]% m# k Z' b" e- E8 {2 r* H* E0 E+ f K: G1 t5 l
' j/ E2 M1 m% Z1 W2 a5 Q8 ^
+ P7 c$ }. Q" }- b( i1 l2 u8 K% v! c! a6 t$ X
' V3 G. w0 @6 v Y% _
+ V/ R( x3 A' V b1 a6 Y+ T$ s9 r
) |- w5 o% S( \7 L, q5 h' e5 |6 v# S$ B0 ]; R
8 e+ y6 @+ V* K2 v9 _' u2 R+ S* o( m
/ q! ^$ ^9 a9 ?; x
|
|